Tableau と R連携では、計算式からリアルタイムにRのコマンドを(SCRIPT_INTなどを利用して)実行することができます。
しかし、特にリアルタイムで実行する必要がない場合はRの計算結果を埋め込んだデータセットを作成し、結果を含んだファイルに対してTableauから可視化を行うということも可能です。
これによって、計算式にRのコマンドを書かなくても良い、地図連携ができる、Tableau Publicにパブリッシュできるなどのメリットが出ます。
もちろんデータが変わったら、出力結果も再作成する必要はあるのですが、こちらの方法も知っておくと大変便利なので紹介したいと思います。
ここではアイリスのデータと、それを採集した地点(データは架空です)のデータ(IRIS_SAMPLE_DATA_ALL_INPUT)を用います。
+ CSVファイルをRに読み込みます。
data <- read.csv(“C:/tmp/IRIS_SAMPLE_DATA_ALL_INPUT.csv”)
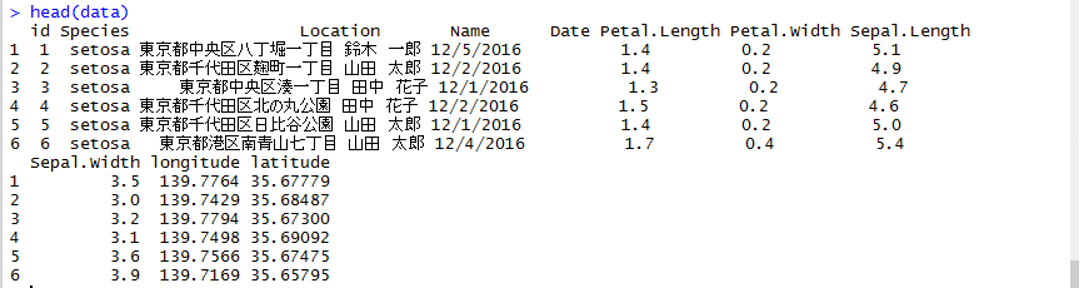
+ 最初の数行を読んで確認します。
> head(data)
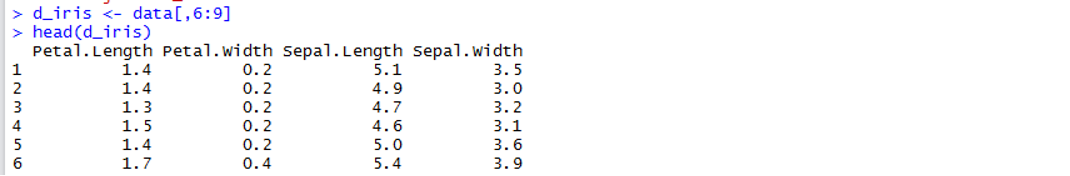
+ クラスタリング分析に利用するアイリスの花の特徴量だけを抜き出すために、
データの5列目から9列目を取り出します。
> d_iris <- data[,6:9]
> head(d_iris)
+ kmeansでクラスタリングを行います。
> cluster <- kmeans(d_iris,3)
> cluster
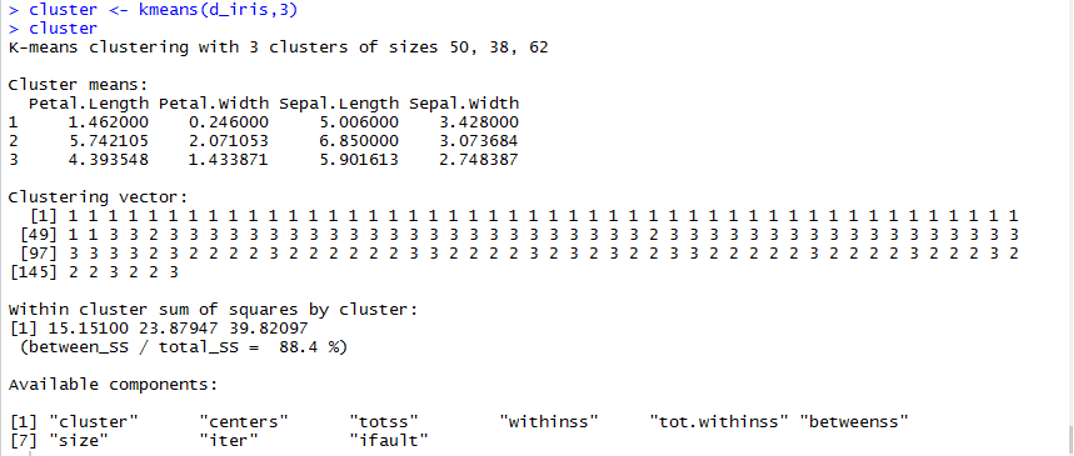
Within cluster sum of squares by cluster:
+ クラスタリングされた結果をclに格納します。
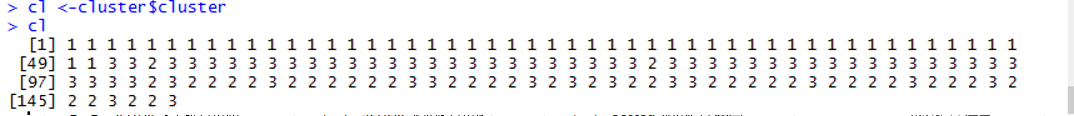
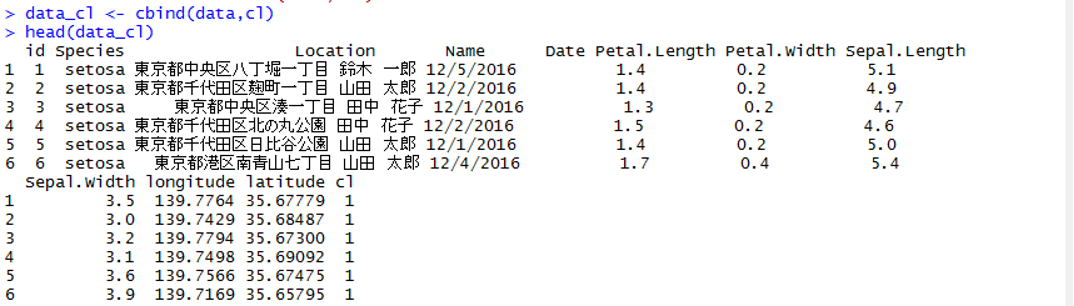
+ cbind使って元のデータと結合します。
> data_cl <- cbind(data,cl)
> head(data_cl)
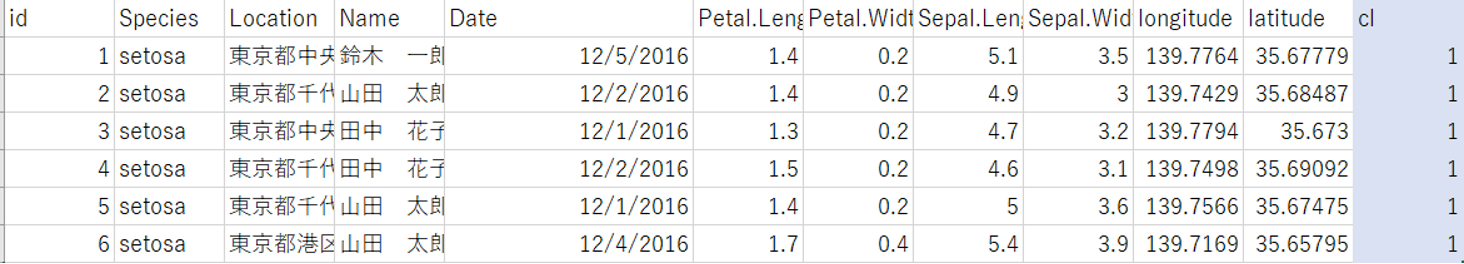
> write.csv(data_cl,”C:/tmp/IRIS_DATA_CL.csv”)
+ 元のデータにクラスタリングの結果が追加されていることを確認します。
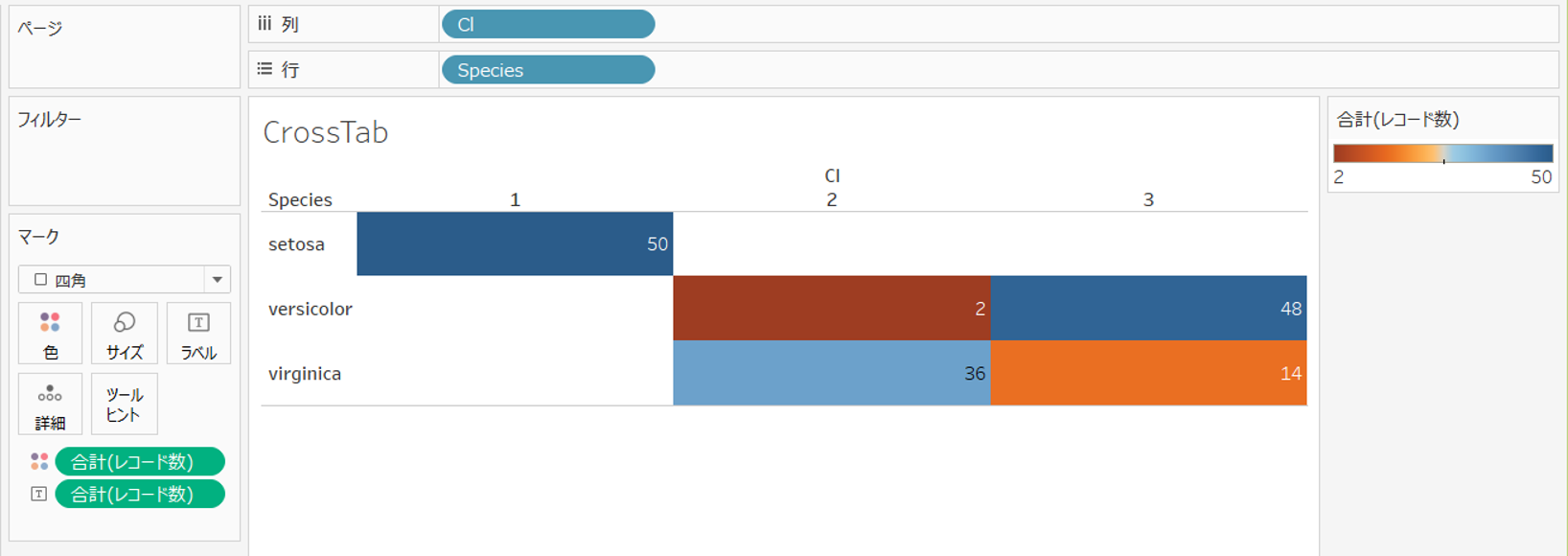
+ Tableauから出力データに接続します。
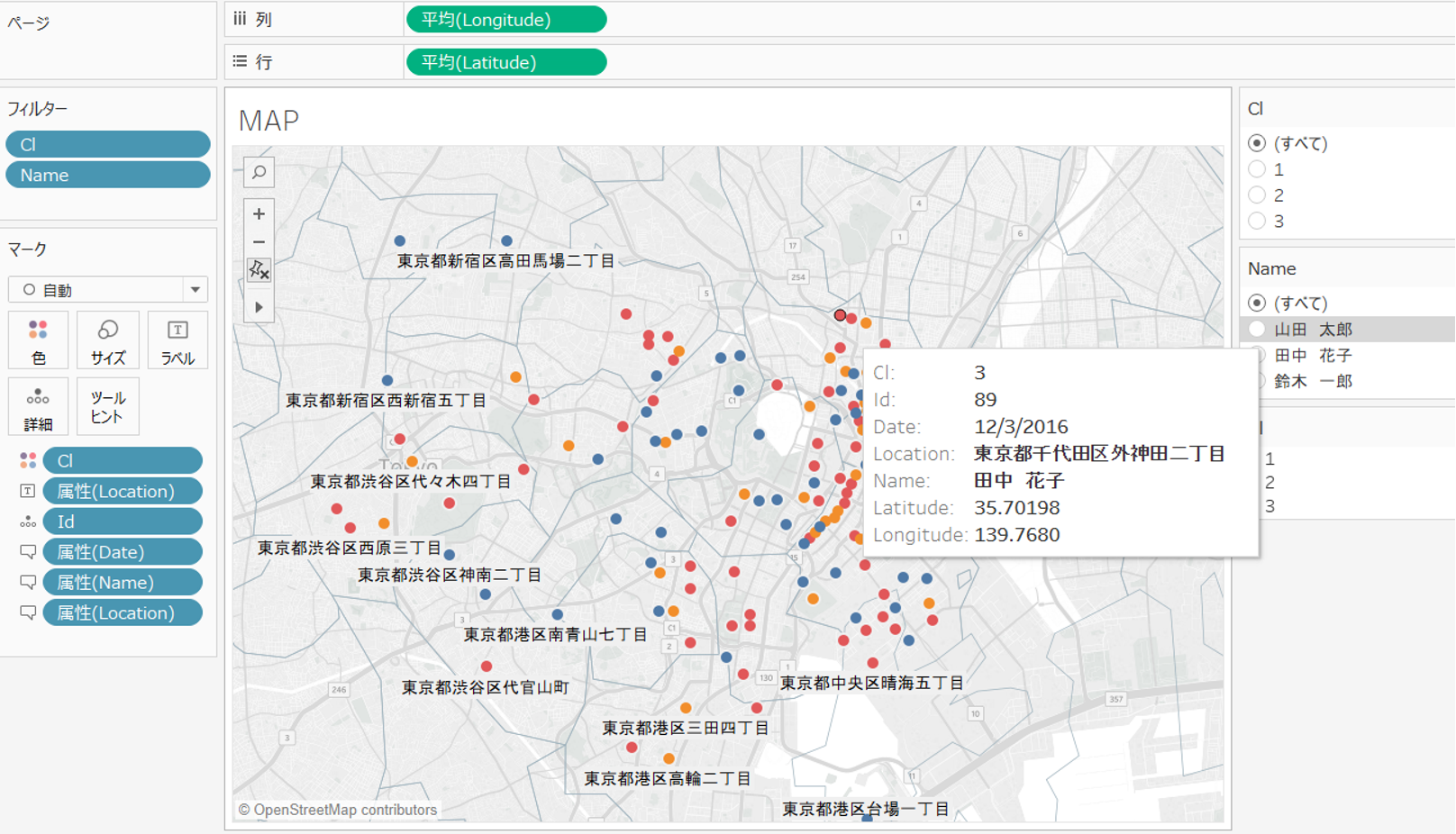
Spicesを行に、Cl(判別されたクラスター)を行に、レコード数をテキストと色にドラッグすると、実際の種別とクラスターによる判別の差異が可視化できます。
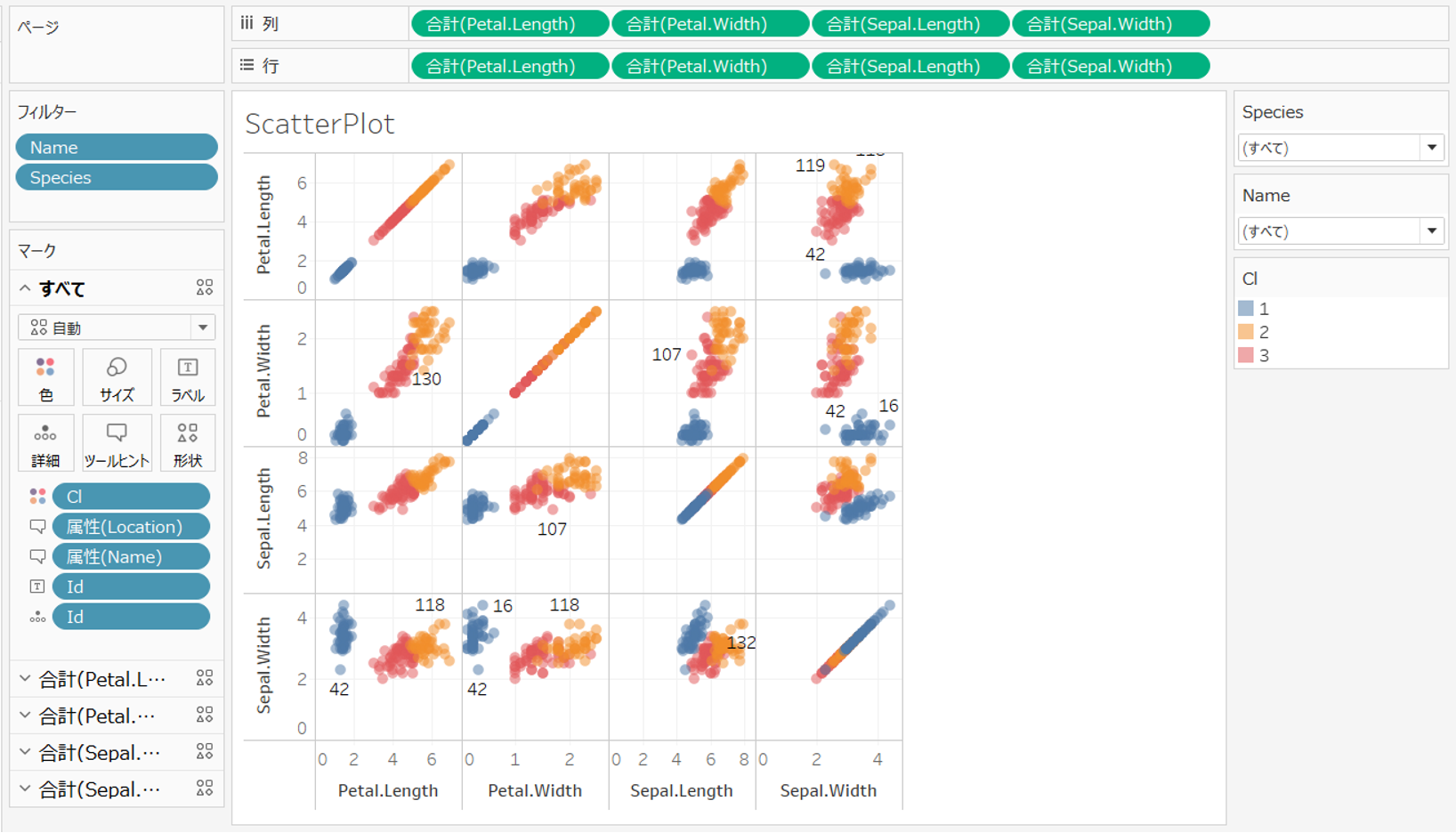
+ 散布図として可視化することも可能です。
+ マップにプロットすると採取地がプロットできます。ツールチップに採取日や採取者の情報を入れることができるので、確認したいプロットにマウスをあてることで詳細情報が簡単に確認できます。